Postado em 6/07/2020
Conteúdo
- Classificação de Vinhos
- Conjunto de Dados de Vinhos
- Verificando a correlação entre as colunas
- Criando os Modelos de Treinamento
- Considerações Finais
Classificação de Vinhos
Escolher um bom vinho não é uma tarefa fácil. Até mesmo os grandes especialistas levam horas para escolher uma garrafa. De fato, são tantas informações que os rótulos dos vinhos possuem que a tarefa de selecionar um bom vinho para acompanhar o seu jantar ou para presentear um(a) amigo(a) torna-se uma odisséia.
Tipo de uva, graduação alcoólica, acidez cítrica, resíduos de açúcar e cloretos são alguns dos itens que podem ser extraídos dos vinhos e que por si só já são capazes de dar um nó na cabeça de qualquer pessoa. É claro que não utilizamos todos esses atributos para selecionar um vinho, sendo eles utilizados para a tarefa de classificação ou regressão utilizando aprendizagem de máquina.
Para esse exemplo, primeiramente iremos importar as bibliotecas básicas que serão utilizadas nesse exemplo.
import pandas as pd
import numpy as np
import sklearnAlém das bibliotecas Pandas e NumPy, também iremos utilizar o Scikit-learn para utilizar os modelos de aprendizagem Random Forest, Gradiente Descendente Estocástico e Support Vector Machine.
Conjunto de Dados de Vinhos
O conjunto de dados de vinhos possui dois subconjuntos de dados que estão relacionados a variantes de vinho tinto e vinho branco português. Esses conjuntos de dados podem ser vistos como tarefas de classificação ou regressão. As classes são ordenadas e não balanceadas (por exemplo, há muito mais vinhos normais do que excelentes ou ruins).
As características químico-físico dos vinhos possuem 11 categorias. A variável de saída possui uma nota de 0 a 10 que é um valor médio atribuído por 3 especialistas de vinhos de cada uma das variantes de vinho tinto e vinho branco.
Os atributos dos vinhos são:
Variáveis de entrada (baseadas em testes físico-químicos):
- fixed acidity
- volatile acidity
- citric acid
- residual sugar
- chlorides
- free sulfur dioxide
- total sulfur dioxide
- density
- pH
- sulphates
- alcohol
Variável de saída (com base em dados sensoriais):
- quality (pontuação entre 0 e 10)
Dataset disponível em: https://archive.ics.uci.edu/ml/datasets/Wine+Quality
data = pd.read_csv('winequality-red.csv', delimiter=';')
df.head()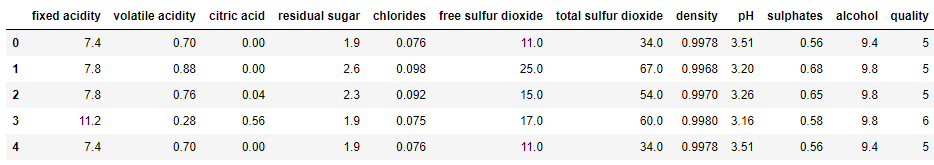
Verificando a correlação entre as colunas
Como esse conjunto de dados possui muitas características dos vinhos, uma boa abordagem inicial é verificar a correlação entre os valores das colunas (entradas) com a classe (saída).
col_list = ['fixed acidity','volatile acidity','citric acid','residual sugar','chlorides','free sulfur dioxide','total sulfur dioxide','density','pH','sulphates','alcohol']
for x in col_list:
corr =data[x].corr(data['quality'])
print (x, corr)A saída da correlação apresentou os seguintes valores:
fixed acidity 0.12405164911322425
volatile acidity -0.39055778026400717
citric acid 0.22637251431804145
residual sugar 0.013731637340066266
chlorides -0.12890655993005273
free sulfur dioxide -0.05065605724427635
total sulfur dioxide -0.18510028892653782
density -0.1749192277833501
pH -0.05773139120538212
sulphates 0.2513970790692613
alcohol 0.476166324001136
A príncipio podemos verificar que existem 3 atributos com valores negativos altos, sendo eles acidez volátil, sulfato de dioxido livre e pH. Esses valores podem indicar que os atributos não possuem tanta relevância para a classe de saída, porém iremos explorar melhor esses atributos mais a frente. Para uma melhor visualização, vamos utilizar as bibliotecas Matplotlib e Seaborn.
import matplotlib.pyplot as plt
import seaborn as snsE então plotar um mapa de calor mostrando a correlação entre os atributos:
corr = data.corr()
fig = plt.figure()
fig, ax = plt.subplots(figsize =(15,10))
g = sns.heatmap(corr, ax=ax, cmap=plt.cm.Blues, annot=True)
ax.set_title('Correlação entre os atributos')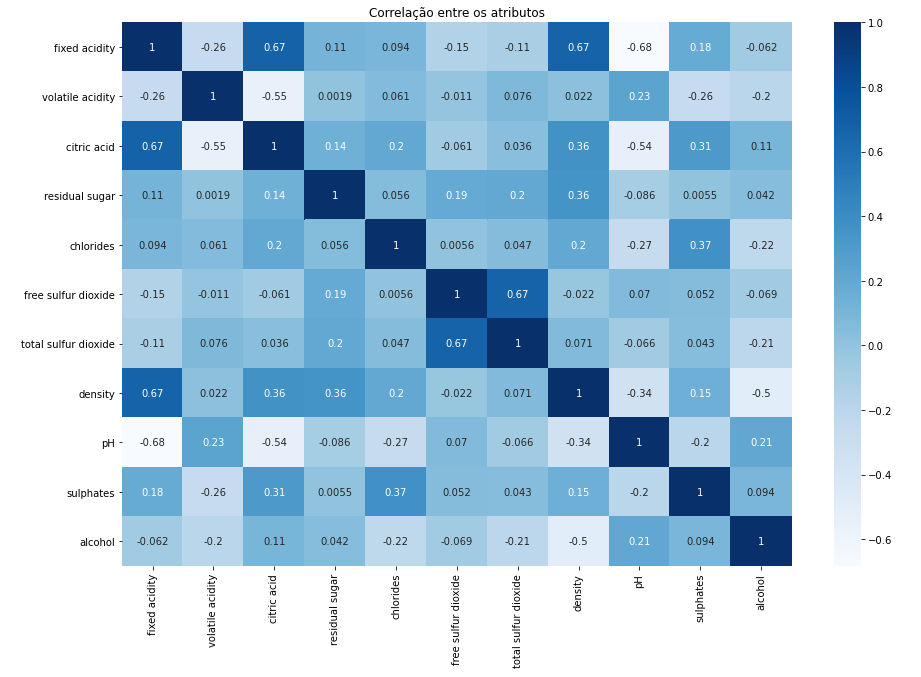
Com a visualização do mapa de calor mostrando a relação entre todos os atributos e a classe de saída, podemos ter uma melhor representação da importância de alguns atributos para a determinação da qualidade dos vinhos. Os atributos mais próximos da cor branca podem ser considerados fracos enquanto que os que estiverem com a cor azul mais forte, podem ser considerados importantes.
Vamos verificar a relação de um dos atributos com a qualidade. Para isso, usaremos um gráfico de barras.
fig = plt.figure(figsize=(10,6))
sns.barplot(x='quality', y='fixed acidity', data=data)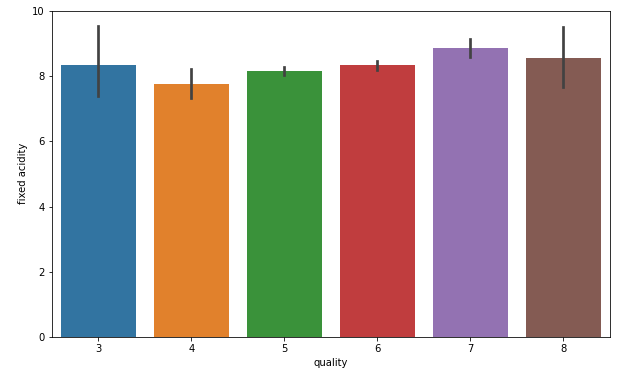
De fato a acidez fixada possui alguma relação com a qualidade dos vinhos, tendo uma distribuição bastante uniforme dentro do conjunto de dados. Podemos fazer outras tipos de análises exploratórias com os dados para identificar outras características importante, porém vamos seguir em frente e construir nosso modelo de classificação de vinhos.
Criando os Modelos de Treinamento
Nosso objetivo é fazer a classificação binária para a variável de destino, que em nosso caso é a qualidade do vinho. Primeiro, iremos dividir a qualidade do vinho como boa ou ruim com base em um limite.
bins =(2, 6, 8)
group_names = ['bad', 'good']
data['quality'] = pd.cut(data['quality'], bins=bins, labels=group_names)Em seguida, vamos excluir a coluna com as classes.
X = data.drop(columns='quality', axis=0)
y = data['quality']E o próximo passo é dividir o conjunto de dados em treinamento e teste. Iremos aplicar o Princípio de Pareto, e usar 20% para treino e 80% teste.
from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=42)Uma última modificação que faremos com os dados será padronizar a escala dos atributos. Essa normalização ajudará a melhorar o desempenho dos modelos ou seja, colocar os todos os dados na mesma escala. Quando não fazemos esse tipo de processamento de dados, alguns algoritmos podem ter um desempenho ruim e gerar modelos ineficientes. Como iremos construir 3 modelos diferentes, esse passo é muito importante.
from sklearn.preprocessing import StandardScaler
sc = StandardScaler()
X_train = sc.fit_transform(X_train)
X_test = sc.fit_transform(X_test)O primeiro modelo será com Random Forest:
from sklearn.ensemble import RandomForestClassifier
rnd = RandomForestClassifier()
rnd.fit(X_train, y_train)
y_pred = rnd.predict(X_test)
from sklearn.metrics import classification_report
print (classification_report(y_test,y_pred))E o resultado obtido:
precision recall f1-score support
bad 0.90 0.97 0.93 273
good 0.71 0.36 0.48 47
accuracy 0.88 320
macro avg 0.80 0.67 0.71 320
weighted avg 0.87 0.88 0.87 320
Nada mal. Foi obtido 88% de precisão. Vejamos o modelo com Gradiente Descendente Estocástico:
from sklearn.linear_model import SGDClassifier
sgd =SGDClassifier(penalty='None')
sgd.fit(X_train, y_train)
y_pred =sgd.predict(X_test)
print (classification_report(y_test, y_pred))E o resultado obtido:
precision recall f1-score support
bad 0.91 0.91 0.91 273
good 0.48 0.49 0.48 47
accuracy 0.85 320
macro avg 0.70 0.70 0.70 320
weighted avg 0.85 0.85 0.85 320
Com com Gradiente Descendente Estocástico foi obtido 85% de precisão. Vejamos o último modelo proposto com SVM:
from sklearn import svm
svc = svm.SVC()
svc.fit(X_train, y_train)
y_pred = svc.predict(X_test)
print (classification_report(y_test, y_pred))E o resultado obtido:
precision recall f1-score support
bad 0.88 0.98 0.93 273
good 0.71 0.26 0.37 47
accuracy 0.88 320
macro avg 0.80 0.62 0.65 320
weighted avg 0.86 0.88 0.85 320
E finalmente, o modelo SVM tamb+em obteve 88% de precisão.
Considerações Finais
Nesse tutorial utilizamos 3 modelos diferentes para classificar vinhos baseados em seus atributos físicos-químicos. Obtivemos bons resultados de precisão 88% utilizando Random Forest e SVM. Esses modelos podem ser aplicados a outras bases disponibilizadas na Internet, bastando fazer alguns ajustes quando necessário. Todo o código e mais um pouco está disponível no meu GitHub.
Os passos de execução deste tutorial foram testados com Python 3.6 e tudo ocorreu sem problemas. No entanto, é possível que alguém encontre alguma dificuldade ou erro no meio do caminho. Se for o caso, por favor comente a sua dificuldade ou erro neste post.